|
 |
||
|
|
|
||
|
|
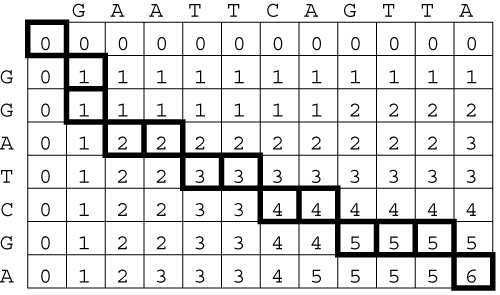
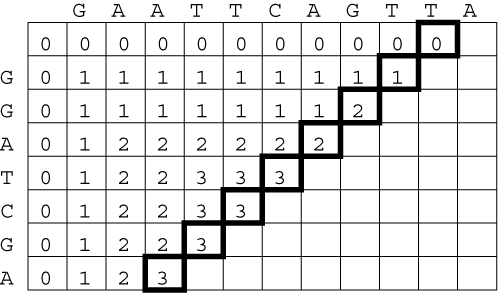
De nos jours, la comparaison et l'alignement de caractères provenant d'un alphabet fini est une tâche fondamentale dans plusieurs domaines d'application, allant de l'examen d'un texte complet à l'analyse informatique en biologie. La comparaison de séquences s'avère plus particulièrement indispensable en biologie moléculaire. Les fragments d'ADN et les protéines peuvent, en effet, être représentés tous deux sous forme de chaînes de caractères (issus d'alphabets de 4 et de 20 symboles, respectivement) et la similarité des chaînes fournit des informations très utiles quant aux relations fonctionnelles, structurelles et évolutives des molécules correspondantes. Les séquences biologiques peuvent différer en raison de substitutions locales, d'insertions ou de suppressions d'un ou de plusieurs caractères dans chacune des chaînes. La complexité de la comparaison provient du grand nombre de combinaisons possibles entre ces trois mutations de base. On peut évaluer la comparaison entre deux chaînes, soit en terme de distance d'édition, soit en terme de résultat de similarité. La distance d'édition donne la mesure du nombre minimal de mutations nécessaires pour transformer la première chaîne afin d'obtenir la seconde ou, autrement dit, du nombre minimal d'opérations d'édition requises pour disposer de deux chaînes identiques. Le résultat de simularité mesure le nombre maximal d'identités résiduelles entre les deux chaînes. Grâce aux techniques de programmation dynamique, l'évaluation des deux métriques s'effectue en une durée de temps polynomiale.
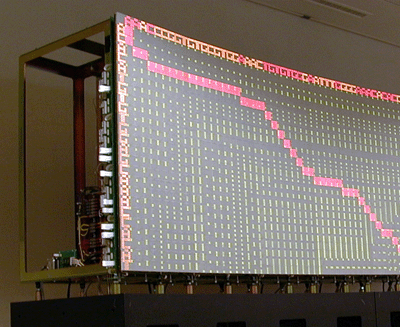
Needleman et Wunsch ont proposé l'algorithme clef pour l'évaluation de similarité entre deux chaînes de longueur N et M. L'exécution complète de cet algorithme s'effectue en O(NxM) pas de temps. Sa structure admet une implémentation parallèle sous forme d'un réseau systolique. L'architecture matérielle parallèle en question permet en particulier d'effectuer la comparaison en O(NxM) pas de temps. Dans cette expérience, on présente une implémentation parallèle de l'algorithme de Needleman-Wunsch sur le BioWall.
La performance en vitesse du BioWall ne soutient pas la comparaison avec les autres implémentations parallèles de l'algorithme de Needleman-Wunsch. Le BioWall souffre, en effet, des limitations temporelles typiques aux plate-formes de prototypage de grande taille. L'implémentation de l'algorithme de Needleman-Wunsch sur le BioWall constitue cependant une conception expérimentale significative dans le domaine du calcul reconfigurable, du fait de l'architecture particulière des circuits mis en oeuvre.
Pour en savoir plus
Ressources
|